RNA origami kan anvendes inden for syntetisk biologi
Med syntetisk biologi stræber man efter at kunne kontrollere biologiske processer og dermed at kunne skabe designerorganismer til en række industrielle, diagnostiske og terapeutiske anvendelser. Forskere ved Aarhus Universitet har udviklet RNA-origami-svampe og CRISPR-baserede regulatorer til avanceret genetisk kontrol af enzymatiske veje i mikroorganismer med det mål at skabe en billigere og mere bæredygtig produktionen af værdifulde biokemikalier

Syntetisk biologi er et felt der har været i stor udvikling og et af dets grundpiller har været udviklingen af værktøjer til at kunne kontroller biologiske processer meget præcist. Disse videnskabelige værktøjer låner principper fra et væld af forskningsfelter, som, når de kombineres, muliggør unikke anvendelser, der potentielt kan have stor betydning for vores moderne samfund. Brugen af moderne RNA-nanoteknologiske opfindelser i biologisk sammenhæng rummer et enormt potentiale. Kroppens celler har nemlig lige det der skal til for at RNA’et kan folde og udtrykkes, dog er det ikke uden udfordringer. Der er nemlig andre betingelser i cellen der gør at det er et barskt miljø for RNA-molekylerne, der i sig selv er ustabile.
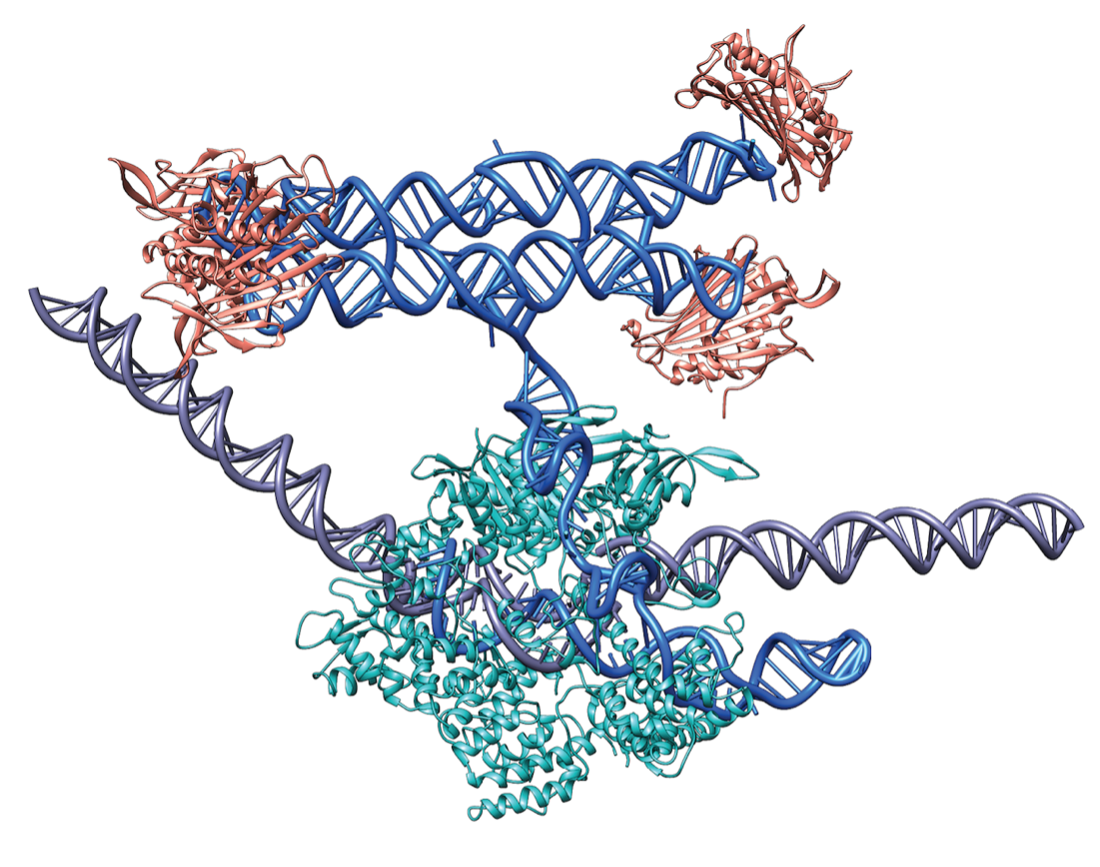
Men i Ebbe S. Andersen’s forskningsgruppe (Andersen Lab) har man udviklet en metode til at takle disse udfordringer med en nyere strukturel RNA-designtilgang, kaldet 'RNA-origami'. Her forsøger man at bygge komplekse menneskeskabte RNA-baserede strukturer, der er stabile i celler. Disse origami-strukturer kan interagere med andre biomolekyler, herunder andre RNA-molekyler og proteiner. Dette giver anledning til helt unikke muligheder for anvendelse, især i forbindelse med genregulering. Dette har forskningsgruppen vist på to forskellige måder, som de for nyligt har offentliggjort i videnskabelige artikler. RNA-origami er en sofistikeret RNA-designplatform, der, når den anvendes i celler, kan generere unikke molekyler til syntetisk biologi-baseret regulering af gener.
RNA-svampe regulerer enzymproduktionen i bakterier
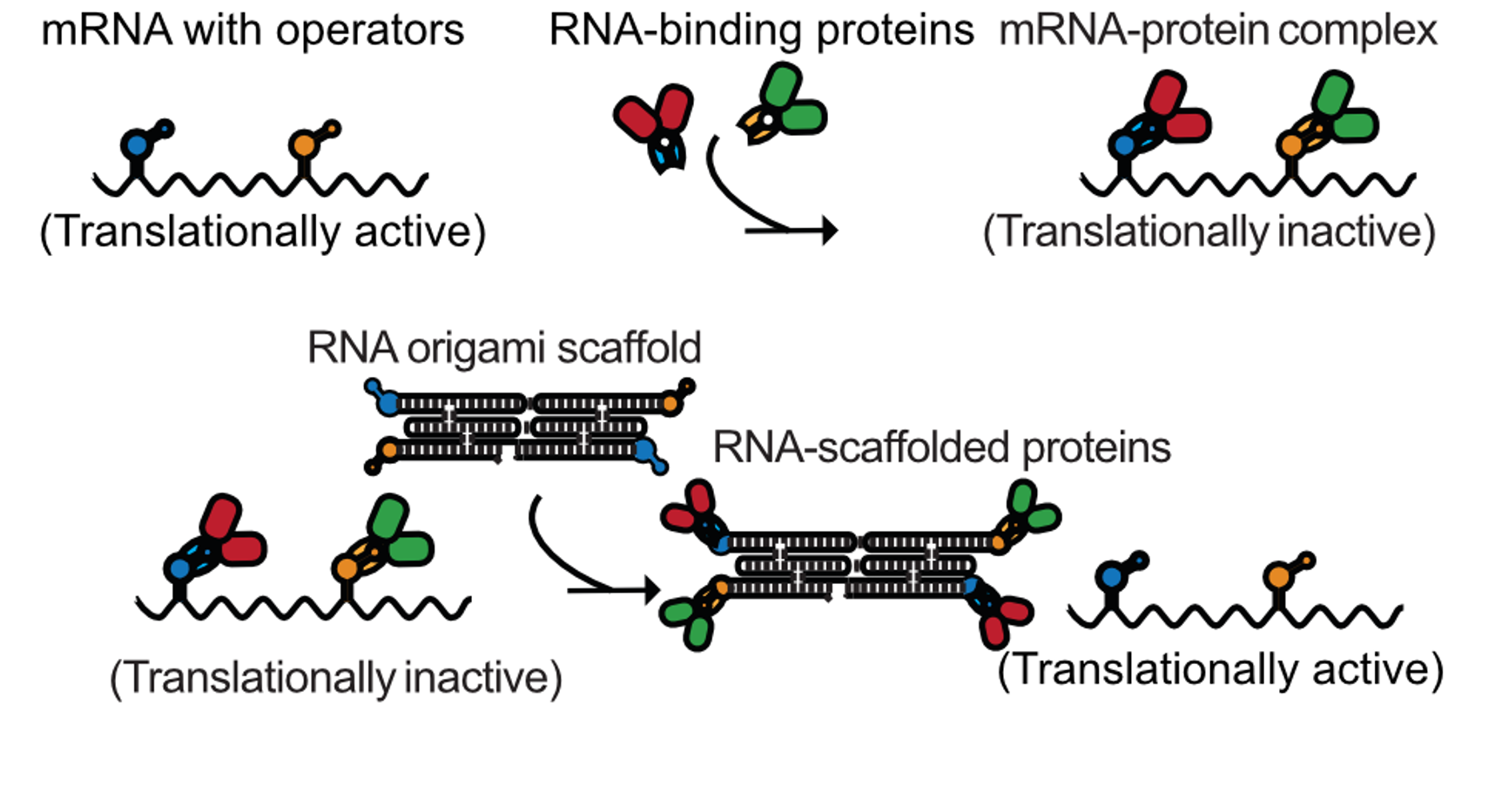
Andersen Lab har bl.a. brugt RNA-origamien til at opnå præcis kontrol af niveauet af proteinproduktionen i bakterier. Dette gjorde de ved at indsætte genstykker i genet for et protein, som fik protein til at binde til sit eget gen og dermed hæmme produktionen af flere af sin egen slags. Bagefter blev RNA-origami dekoreret med et kæmpe antal af de samme proteinbindingssteder. På denne måde fungerer RNA-origamien som en protein-svamp, der indfanger proteiner i cellen, og dermed muliggør ekspression af det ellers selvhæmmede protein. Forskerne kunne derudover vise at de kunne regulere flere proteiner samtidigt og aktivere enzymatiske veje der gjorde at produktionen af bestemte proteiner øgedes.
CRISPR-baserede regulatorer til gærkemiske fabrikker
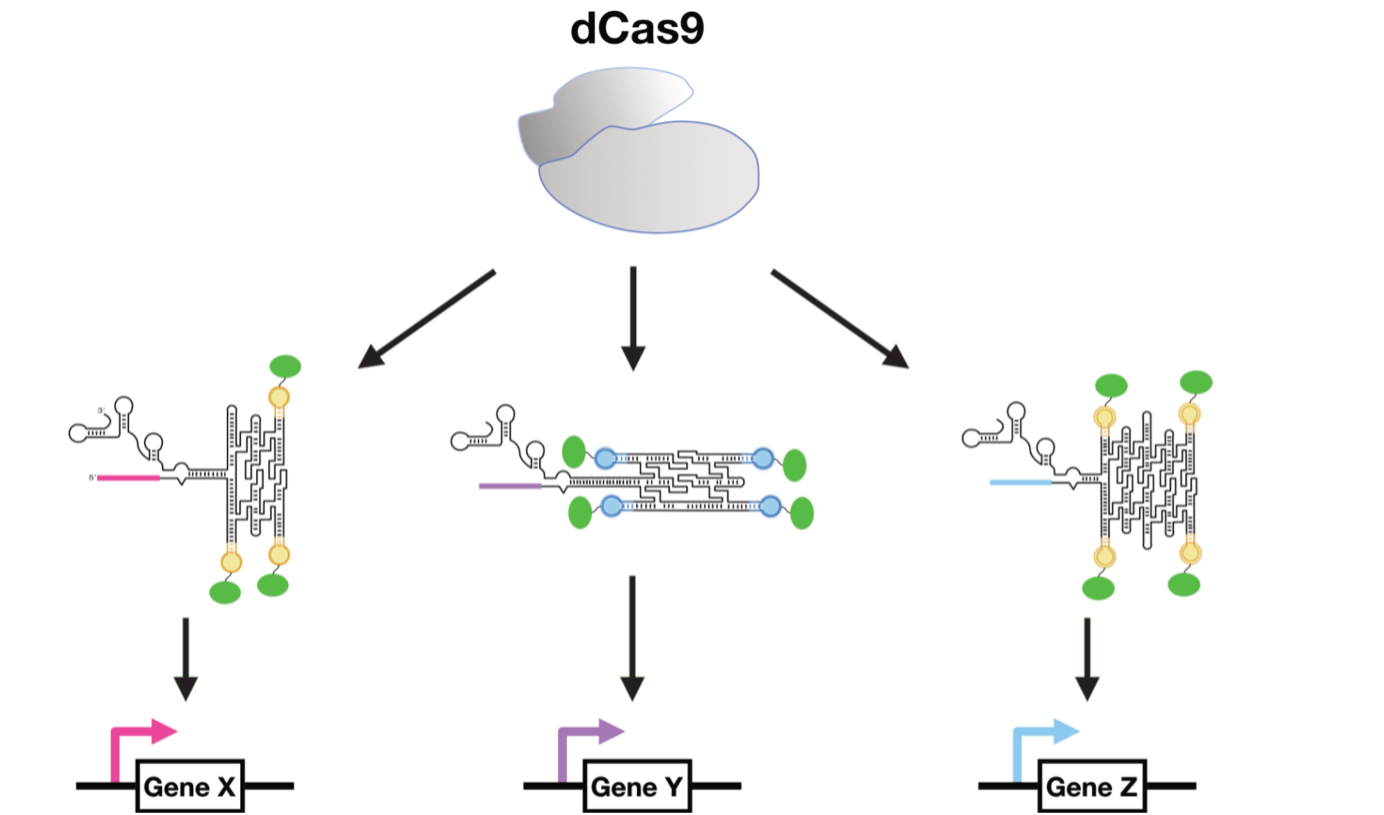
Ved at kombinere RNA-origami med CRISPR, en af de mest populære moderne molekylærbiologiske teknikker, kan de også regulere genekspression i gærceller. RNA-origamierne blev integreret i de små RNA'er, der guider CRISPR-Cas9 til at angribe specifikke sekvenser i DNA-genomet. RNA-origami’erne blev dekoreret med proteinbindingssteder, der var i stand til at rekruttere transkriptionsfaktorer, der kan tænde og slukke for gener. Ved at lade RNA-origami’erne angribe relevante genregioner så førte det til at transkriptionsfaktorerne aktiverede genekspression. Det blev endda vist, at niveauet af ekspression kunne indstilles afhængigt af hvordan RNA-origami’erne vendte samt mængden af rekrutterede transkriptionsfaktorer.
Sidst men ikke mindst kunne de, vha. særlige gærceller som udtrykker anti-cancer-lægemidlet violacein, vise at RNA-origami’erne kunne kontrollere flere enzym-veje på en gang, hvilket førte til øget produktion af dette vigtige biokemialie.
Netop disse opdagelser er vigtige, da det er med til at vise at ved hjælp af RNA-origami kan mikroorganismer bruges som små fabrikker til billig og bæredygtig produktion at f.eks. medicin.
SUPPLERENDE OPLYSNINGER, HERUNDER KONTAKTOPLYSNINGER
Ekstern finansiering
- European Research Council (RNA ORIGAMI─RNA-protein nanostrukturer til syntetisk biologi, 683305)
- Novo Nordisk Fonden (0060694)
Interessekonflikt
Forskerne erklærer, at der ikke er nogen interessekonflikter.
Link til videnskabelige artikler
- Nguyen, M. T. A., Pothoulakis, G. & Andersen, E. S. Syntetisk translationel regulering af proteinbindende RNA Origami-stilladser. ACS Synth Biol 11, 1710-1718 (2022). https://doi.org/10.1021/acssynbio.1c00608
- Pothoulakis, G., Nguyen, M. T. A. & Andersen, E. S. Udnyttelse af RNA-origami-stilladser i Saccharomyces cerevisiae til dCas9-medieret transkriptionel kontrol. Nucleic Acids Res 50, 7176-7187 (2022). https://doi.org/10.1093/nar/gkac470
Kontakt information
Lektor Ebbe S. Andersen
Interdisciplinært Nanoscience Center (iNANO) & Institut for Molekylærbiologi og Genetik
Aarhus Universitet
E-mail: esa@inano.au.dk
