Større og bedre RNA-stilladser til at organisere proteiner
Forskere fra Aarhus Universitet og Caltech har udviklet en metode til at bygge meget større stilladser af RNA, end man hidtil har troet muligt – om end stadig i nanostørrelse – ved hjælp af RNA-origami. Metoden bygger på ny software, som forskerne har gjort tilgængelig online, så også andre forskere kan bruge den til f.eks. at udvikle biosensorer, nanorobotter og medicin – herunder vacciner.
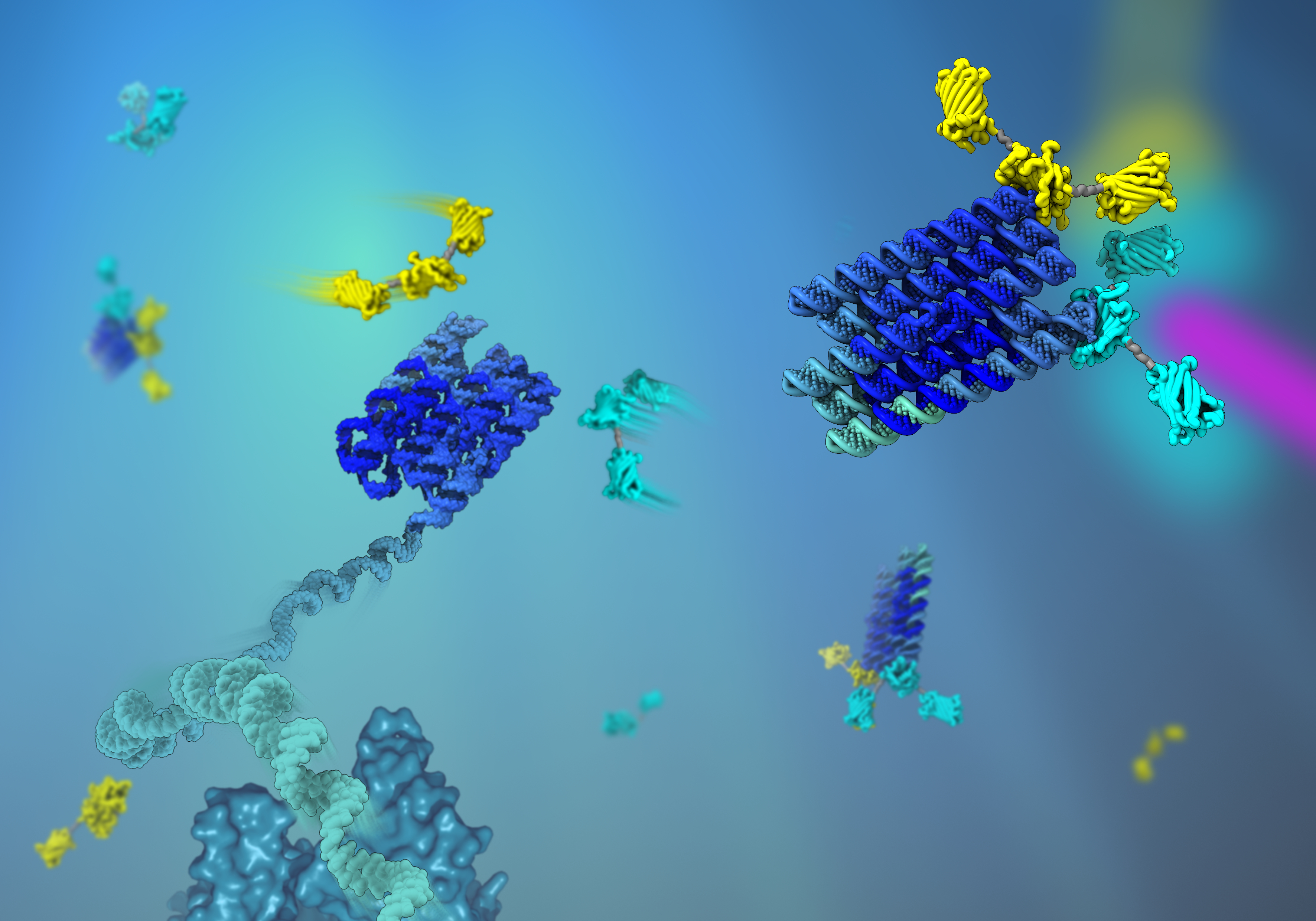
Normalt møder du stilladser, når de bliver brugt som understøttende struktur og midlertidige arbejdsplatforme i forbindelse med byggeri. Men dine egne celler bruger også stilladser, blot i nanostørrelse og af proteiner, til at organisere cellulære processer og til at konstruere din krop. Disse proteinstilladser naturens avancerede stilladser inspirerer bioingeniører til at designe “kunstige” nanostilladser, som kan bruges til at organisere biologiske processer, f.eks. for at optimere biologisk produktion af værdifulde kemikalier.
I et nyt studie publiceret i Nature Chemistry tager forskere fra Aarhus Universitet og California Institute of Technology et stort skridt fremad i rationelt design af RNA-stilladser til at organisere proteiner. Ved at bruge RNA-origami-metoden producerer de meget større stilladser, end man tidligere troede var muligt.
Design-softwaren, der gjorde studiet muligt, har forskerne gjort tilgængeligt online med vejledninger og en dedikeret webserver, så andre forskere kan bygge RNA-stilladser til deres egne formål.
Origami med begrænsninger
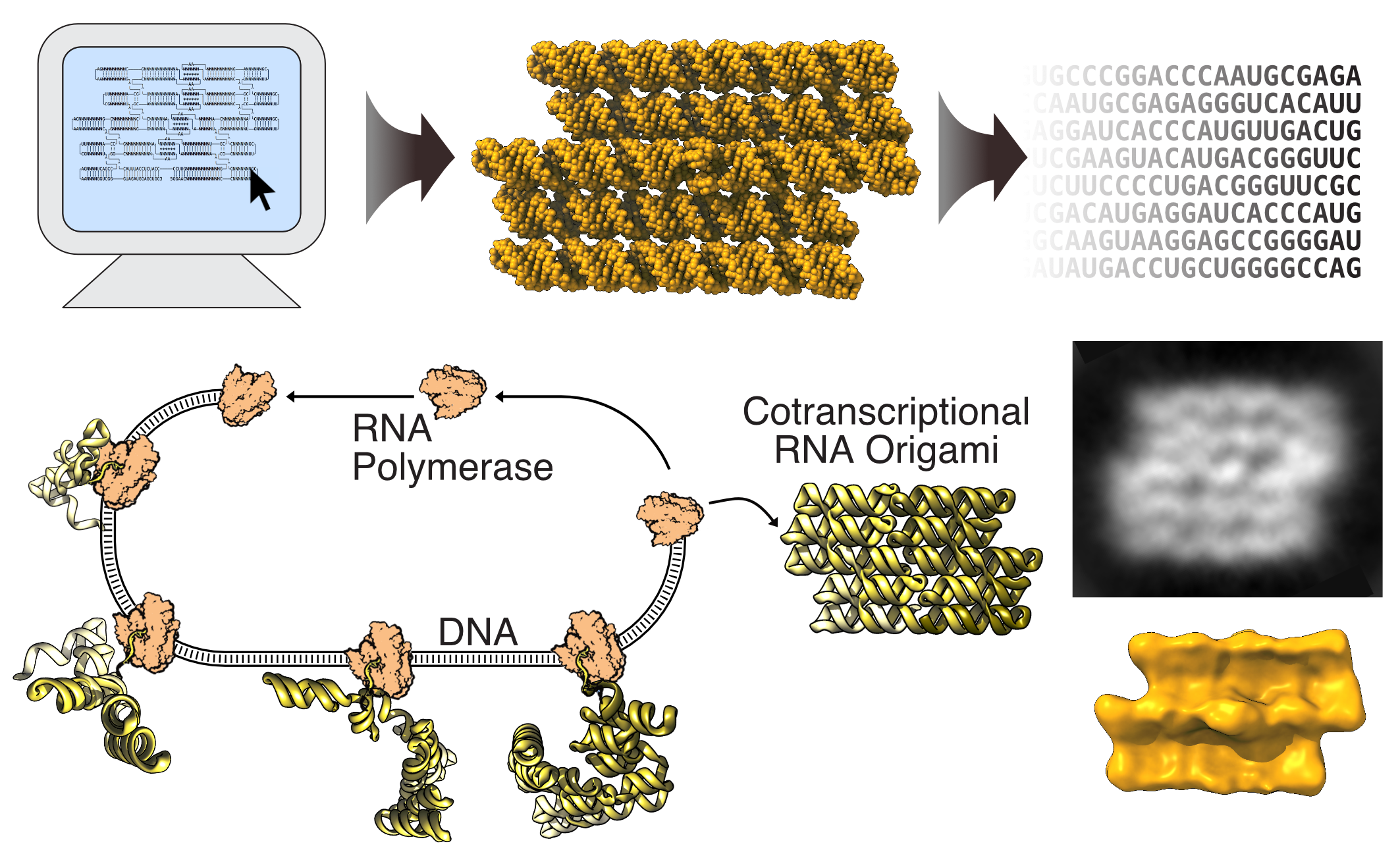
RNA-origami-metoden blev introduceret i 2014 som et byggesæt af RNA-elementer, der kunne kombineres som legoklodser for at konstruere RNA-nanostrukturer, der kan foldes fra en enkelt streng. En vigtig opdagelse var, at disse RNA-strukturer kunne foldes under transskriptionen af en RNA-polymerase; det smart, for det gør det muligt genetisk at kode og udtrykke RNA nanostrukturer i celler.
RNA-origami-metoden havde dog alvorlige begrænsninger:
"I den originale artikel nåede vi en længde på 450 nukleotider for foldet RNA-origami med ganske lavt udbytte. Derudover nåede vi grænserne for RNA-designsoftwaren, der var tilgængelig på det tidspunkt. I den nye artikel løser vi disse begrænsninger,” forklarer Ebbe Sloth Andersen, lektor ved Aarhus Universitet.
Større og bedre RNA-stilladser
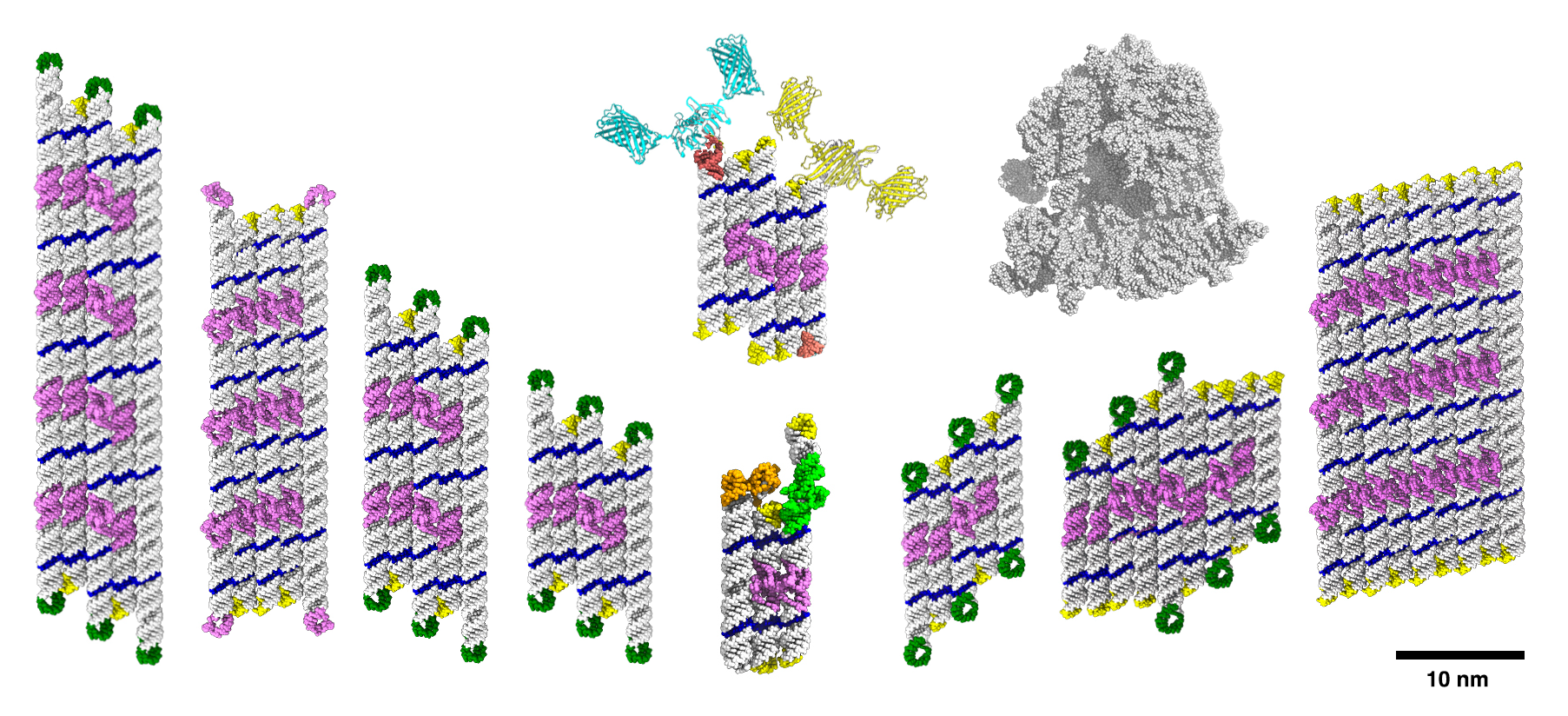
Det aktuelle studie blev muliggjort ved at udvikle ny software, der kan designe et stort panel af RNA-origami-strukturer. RNA-origami-strukturer er designet som flade plader med RNA-dobbeltspiraler, som nu blev udvidet med enten antallet af stablede dobbeltspiraler eller langs spiral-aksen. Eksperimenterne viste, at RNA-origami-strukturer kan skaleres i begge retninger op til en længde på ca. 2000 nukleotider med op til 24 langdistance-interaktioner.
"Vi var overraskede over at kunne producere veldefinerede RNA-strukturer i denne størrelse. Tidligere havde vi kun et begrænset sæt med otte loop-interaktioner at vælge imellem, og det var et stort gennembrud at finde en måde at producere nye sekvenser til disse programmerbare interaktioner," fortæller Cody Geary, adjunkt ved Aarhus Universitet, der udviklede projektet både på Caltech og Aarhus Universitet.
Forskerholdet observerede, at mængden af korrekt foldede origamier faldt væsentligt for de største strukturer. For at forstå hvorfor udførte de et eksperiment, der kunne undersøge effekten af ??forskellige foldestier. Ved at bruge den samme struktur med forskellige foldestier og undersøge udbyttet af foldede strukturer ved elektronmikroskopi, observerede postdoc Ewan McRae foldedefekter i RNA-origami-strukturer.
Derefter brugte de RNA-origami-strukturer med det bedste foldningsudbytte til stilladseksperimenter.
Har aldrig skrevet software før
Den softwarepakke, som gjorde studiet muligt, automatiserer de vigtigste trin i processen med at designe RNA origami. Den består af et bibliotek med scripts, der alle er skrevet af Cody Geary – som aldrig havde skrevet en softwarepakke før.
"Men måske var det en fordel. I sidste ende kører programmet næsten fuldstændigt omvendt i forhold til konventionelle programmer, og løser de sværeste begrænsninger i slutningen. Jeg tror, ??det er hemmeligheden bag, hvad der får det til at fungere så godt", siger Geary.
Designet af RNA origami strukturer starter med konstruktionen af ??et blueprint, der kan sammensættes af foruddefinerede moduler.
- Det første program, kaldet RNAbuild, oversætter tegningen til atommodeller, der giver brugeren mulighed for gradvist at opbygge en RNA-origami-struktur og få feedback på den overordnede form.
- Det andet program, kaldet RNApath, estimerer rækkefølgen af ??foldning og topologiske barrierer, som hjælper brugeren med at evaluere forskellige valg af foldesti.
- Det tredje program, kaldet Revolvr, designer en RNA-sekvens, der kan foldes som det givne blueprint. Revolvr løser mange af de tidligere begrænsninger for RNA-design-algoritmer ved at være i stand til at designe strukturer af stor størrelse og med flere loop-interaktioner med relativt korte beregningstider.
Softwaren giver i løbet af minutter resultater, som ikke kunne opnås på dage eller uger med eksisterende software – der i princippet skulle være i stand til at designe sådanne strukturer, men ikke kan.
Når sekvensen er designet, kan man bestille en DNA-skabelon fra et gensyntesefirma. Med DNA-skabelonen i hånden kan man lave RNA-strukturen med en simpel RNA-transskription. For at se, om RNA-strukturer har dannet sig som forventet, anvendes avancerede mikroskopimetoder til at visualisere strukturen.
Animation af foldningen af en RNA-origami, hvor orange og røde farver angiver potentielle topologiske problemer for foldningsprocessen. Animation: Cody Geary
Fremtidige anvendelser af RNA-origami
Den aktuelle undersøgelse blev udført med rene komponenter i et reagensglas, men RNA-origami kan i princippet udtrykkes i celler. Undersøgelser begynder at dukke op, hvor RNA-origami-strukturer er kodet genetisk og udtrykt i celler eller brugt som medicinske nanopartikler, der kan regulere blodpropper.
"Jeg forestiller mig, at vi vil være i stand til at designe mere og mere komplekse strukturer meget mere frit i tre dimensioner og endda blive i stand til at konstruere bevægelige dele inden for disse strukturer. Denne type fremskridt vil føre til udvikling af mere kompakte designs, der kan fungere som nano-maskiner eller nano-robotter", siger Cody Geary.
Ifølge Ebbe Sloth Andersen ser fremtiden lys ud for anvendelsen af ??RNA-nanostrukturer i både medicin og syntetisk biologi. Med mRNA-vacciner mod coronavirus, der bliver leveret i lipidnanopartikler, er udsigten til levering af medicinske RNA-partikler kommet tættere på virkeligheden, og genetisk udtryk af RNA-origami kan bruges til mange anvendelser inden for syntetisk biologi.
"Vi arbejder nu på at anvende RNA-stilladser som værktøjer inden for bioteknologi. Til dette formål udvikler vi RNA-origami som biosensorer til at detektere metabolitter i celler, som stilladser til regulering af genekspression, og som stilladser til enzymer for at styre metabolisk produktion”, fortæller Ebbe Sloth Andersen.
Supplerende oplysninger | |
| Vi bestræber os på, at alle vores artikler lever op til Danske Universiteters principper for god forskningskommunikation. På den baggrund er artiklen suppleret med følgende oplysninger: | |
| Finansiering | Undersøgelserne har modtaget økonomisk støtte fra det Europæiske Forskningsråd (ERC), National Science Foundation (NSF), Office of Naval Research (ONR), Carlsberg Forskningsfond, Det Frie Forskningsråd (DFF), Natural Sciences and Engineering Research Council of Canada. |
| Læs mere | Link til artiklen i Nature Chemistry Vejledning til RNA-designsoftware og webserver er tilgængelig på https://bion.au.dk/software/rnao-design/ |
| Kontakt | Lektor Ebbe Sloth Andersen. Mail:esa@inano.au.dk Mobil:+45 41178619 Adjunkt Cody Geary. Mail: geary@inano.au.dk Professor Paul Rothemund. Mail:pwkr@dna.caltech.edu. Telefon:+1 626-390-0438 |
